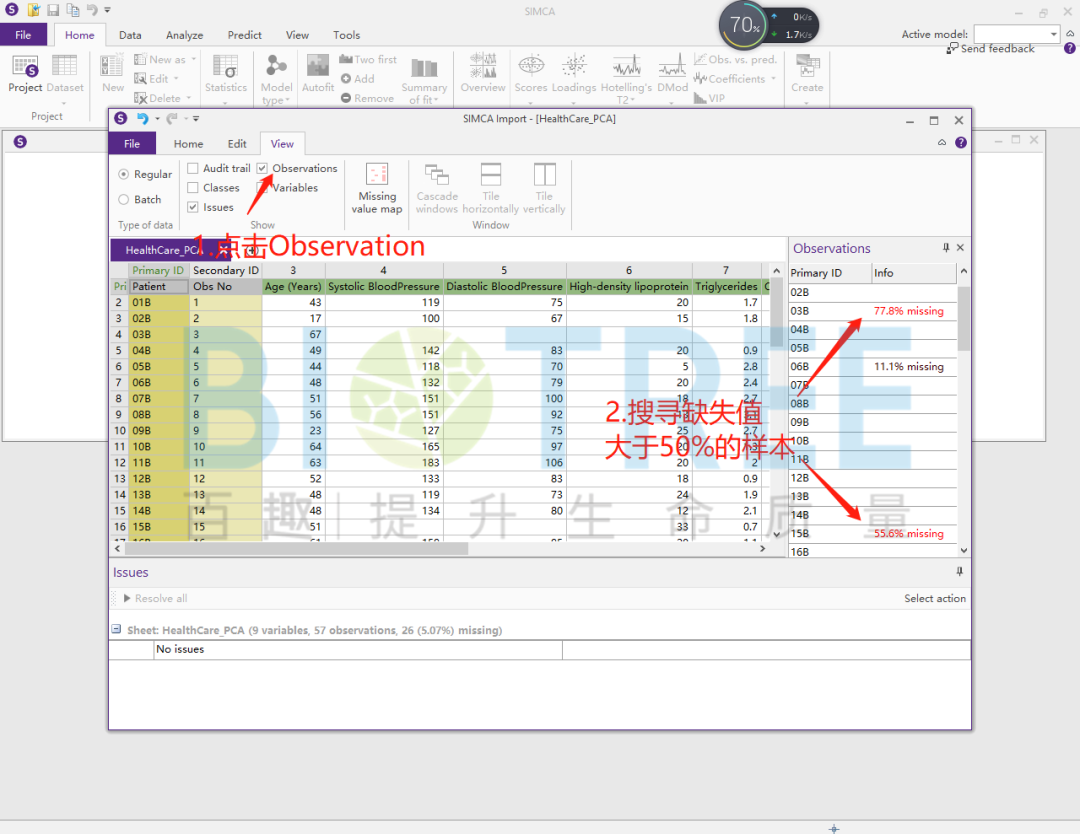
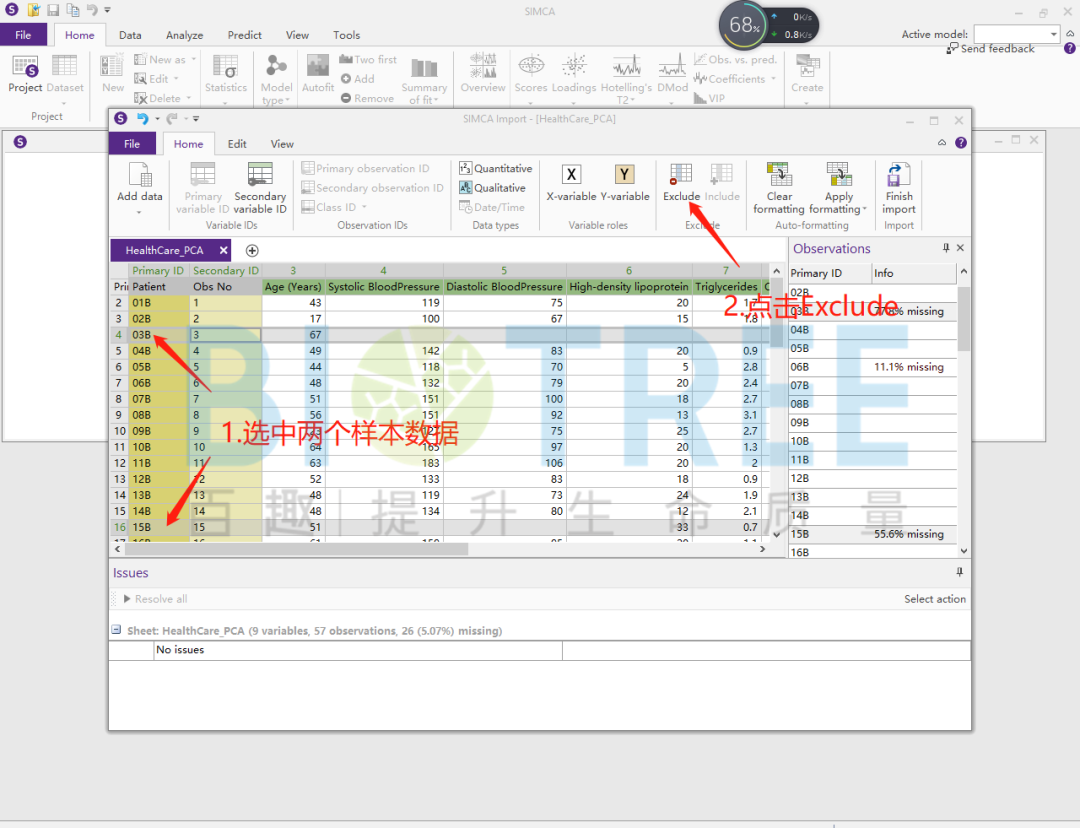
 当样本中的缺失值过多时,样本本身就缺乏了统计学意义,并且极有可能成为异常样本点,因此本案例对缺失值大于50%的样本数据进行去除处理。
当样本中的缺失值过多时,样本本身就缺乏了统计学意义,并且极有可能成为异常样本点,因此本案例对缺失值大于50%的样本数据进行去除处理。
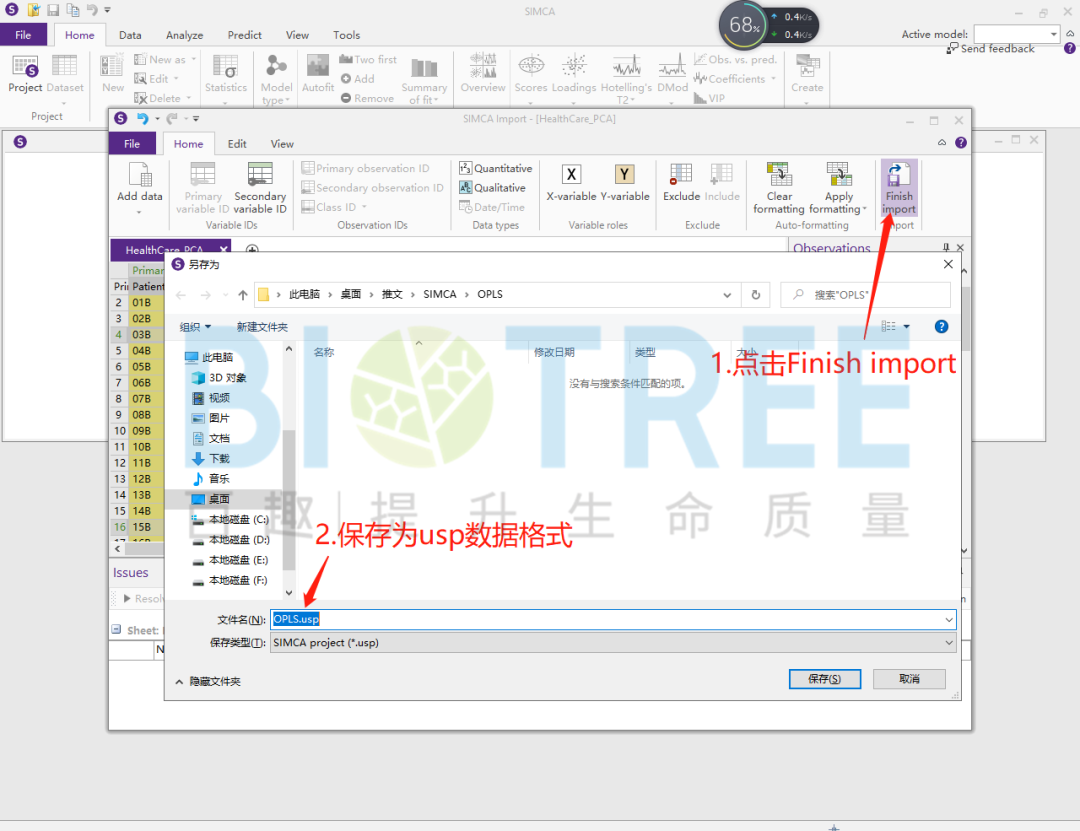
通过OPLS得分图和载荷图相结合的方式,对其进行解读可得到更多的信息。两者相结合可以看出哪些样本与Y变量相关性较强,哪些样本相关性较弱。

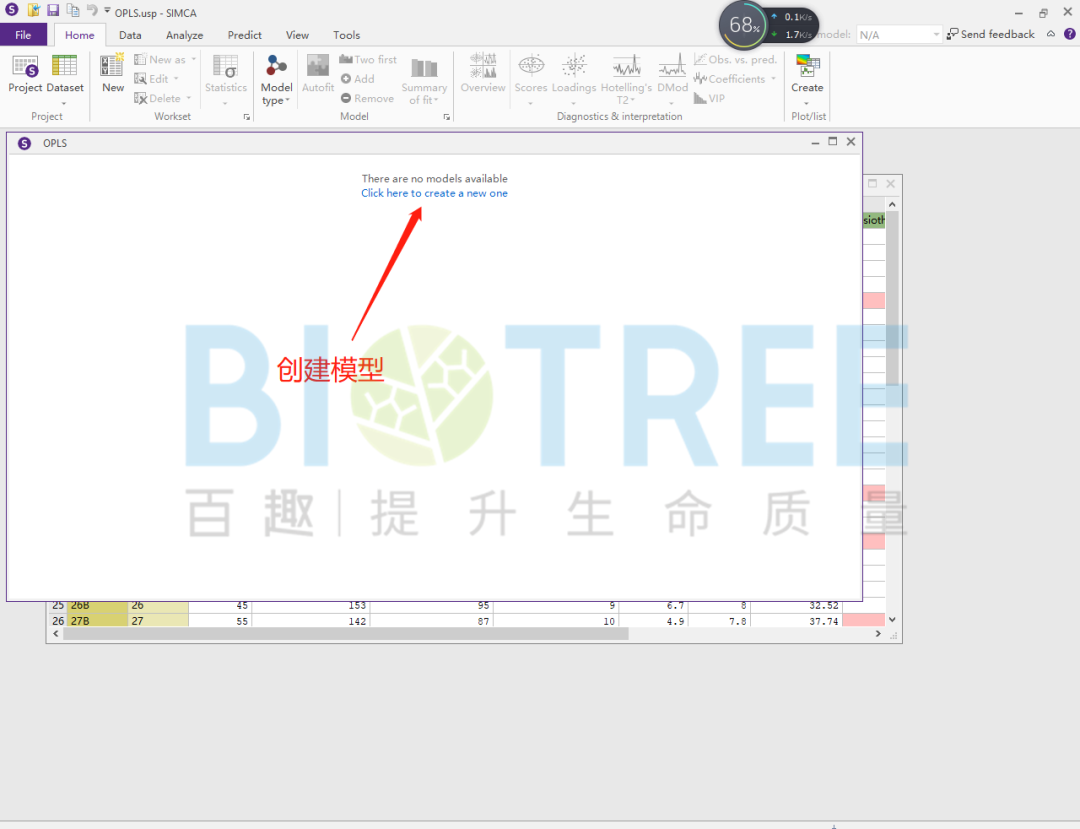
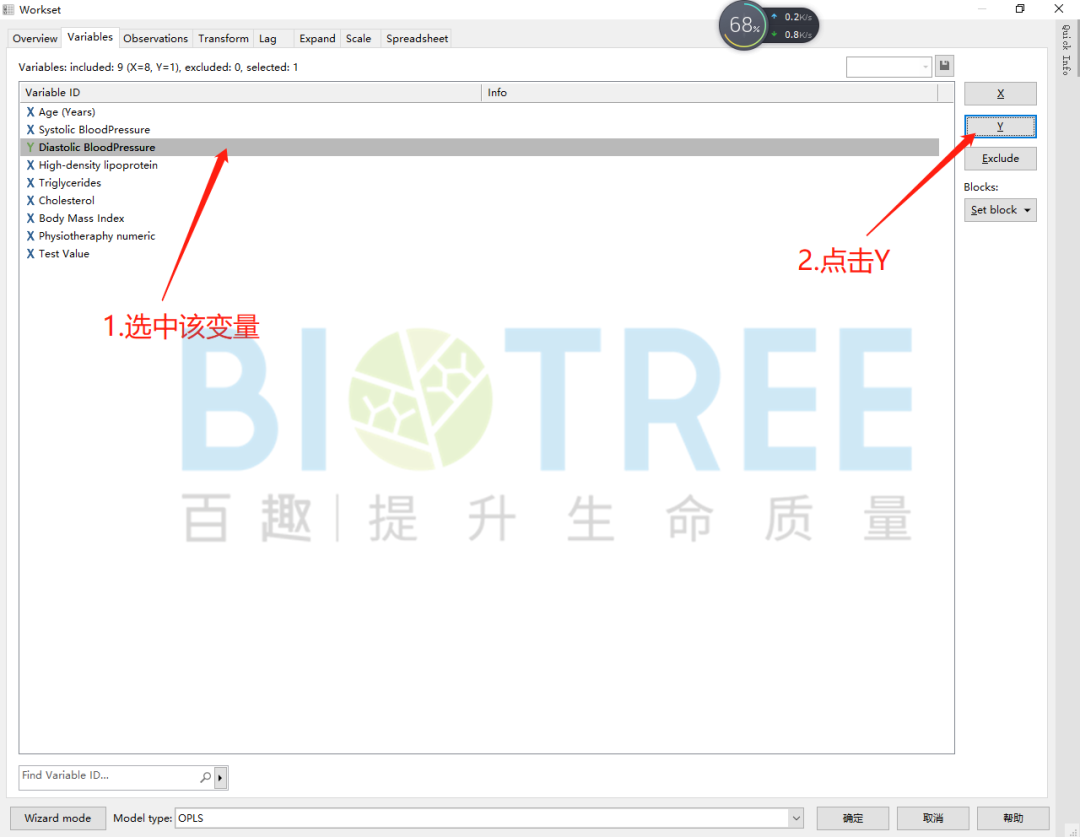
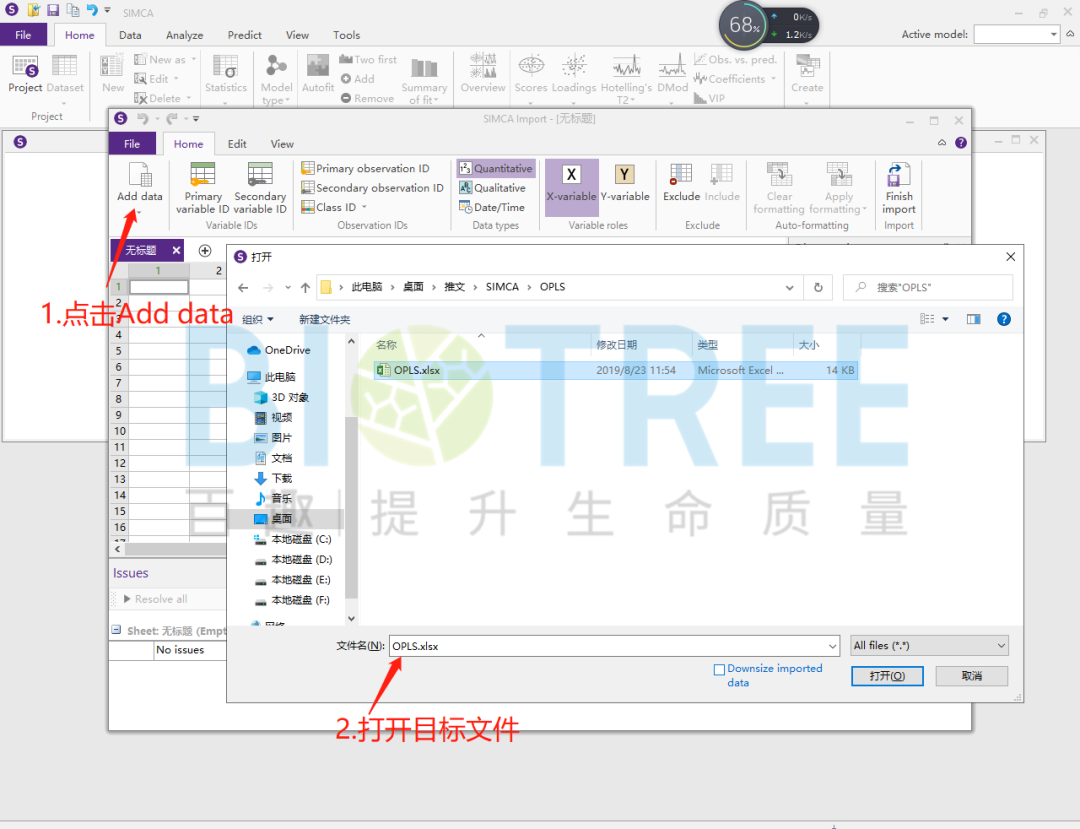
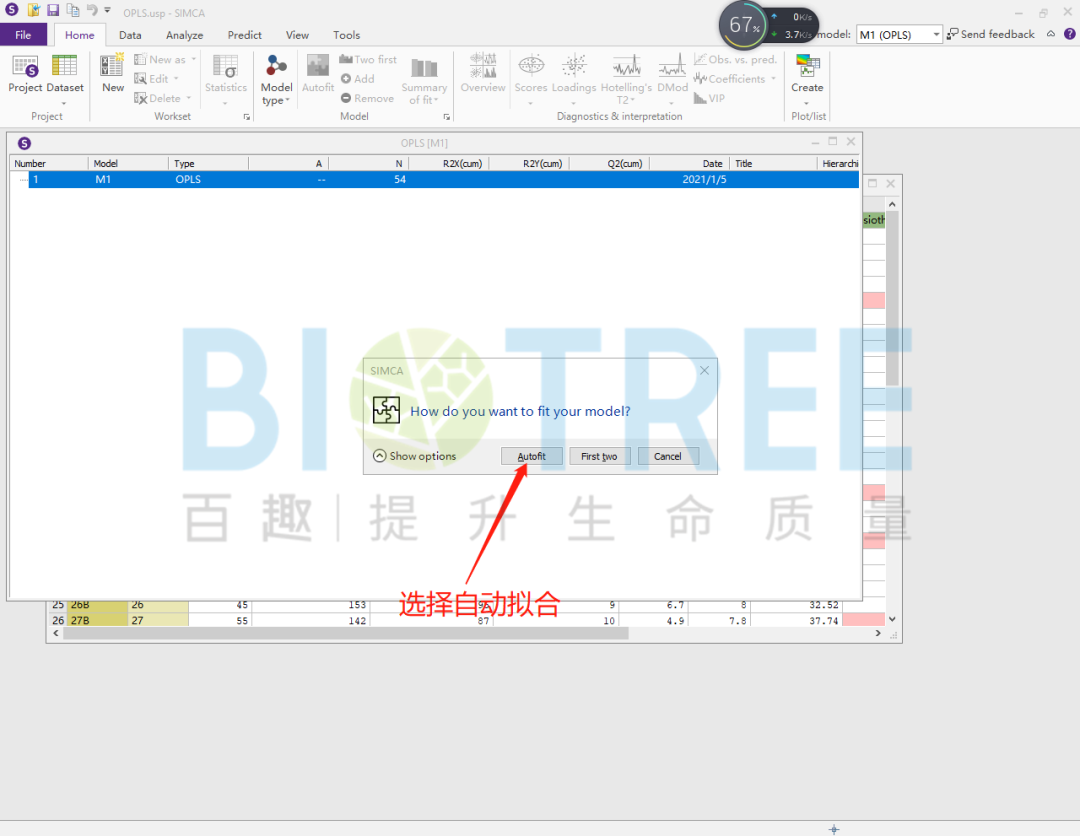
进行OPLS模型的构建首先需要设置Y变量。
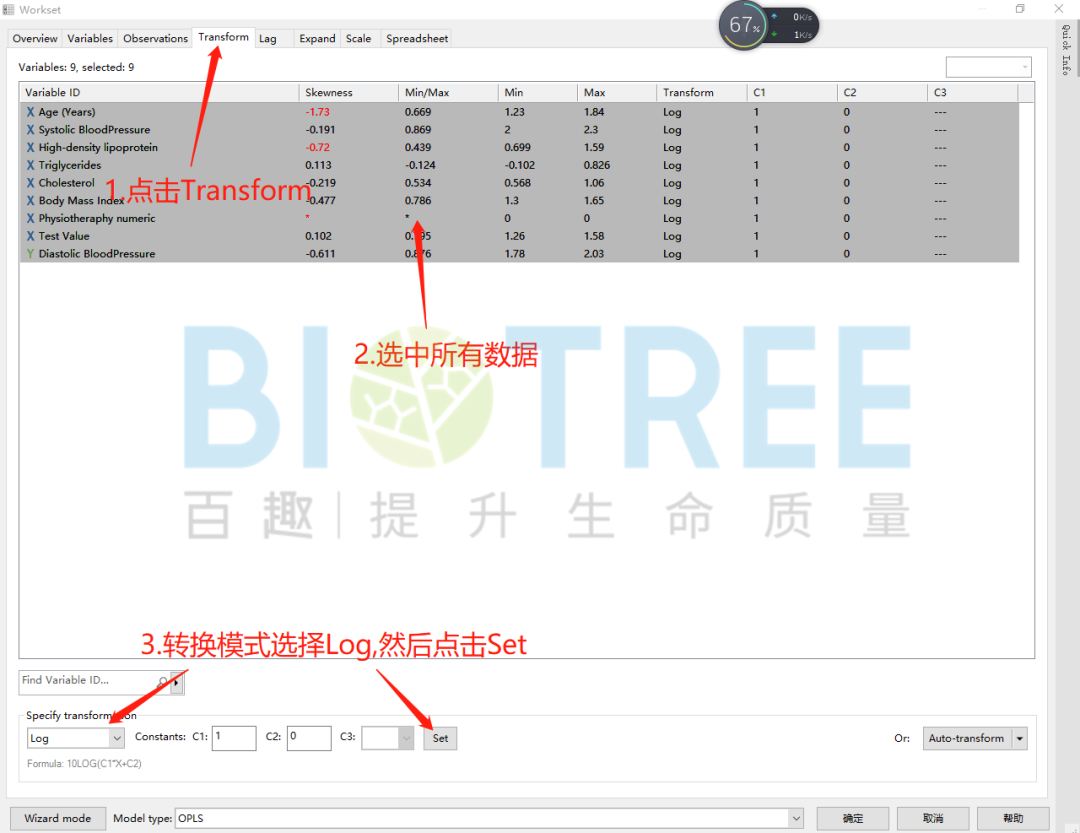
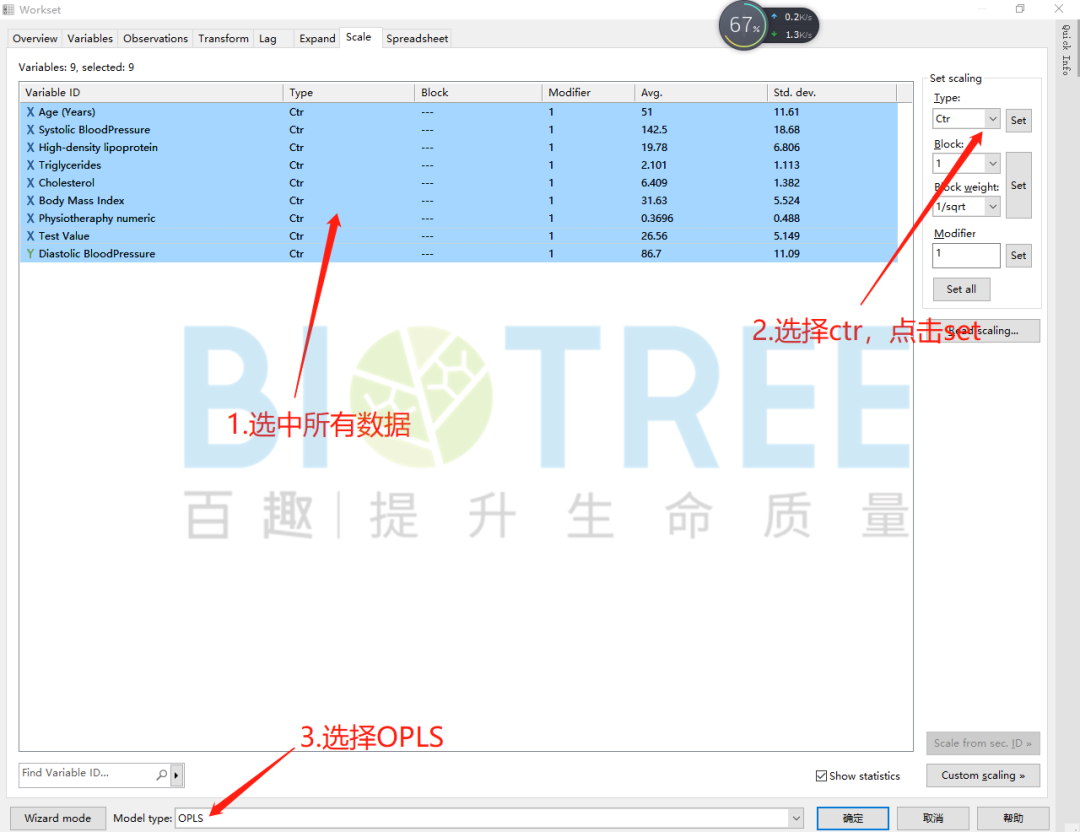
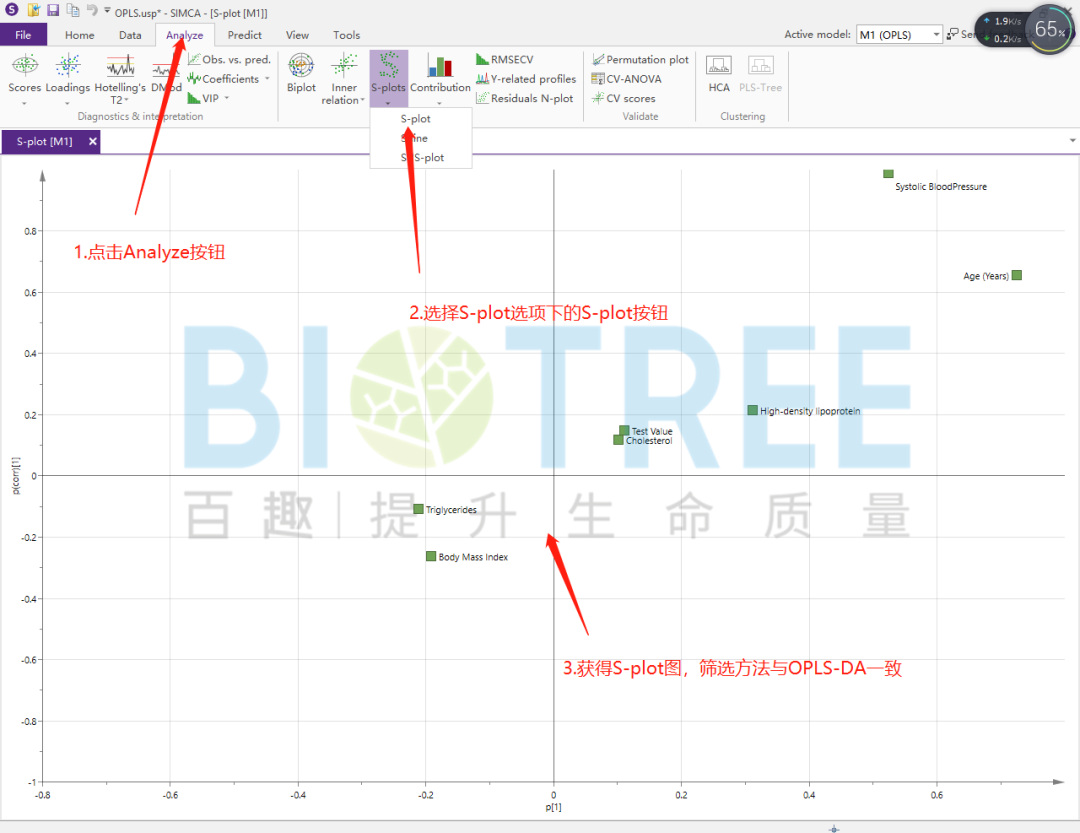
Scale方式选择ctr或par有助于S-plot图形成S形这有利于数据的筛选。
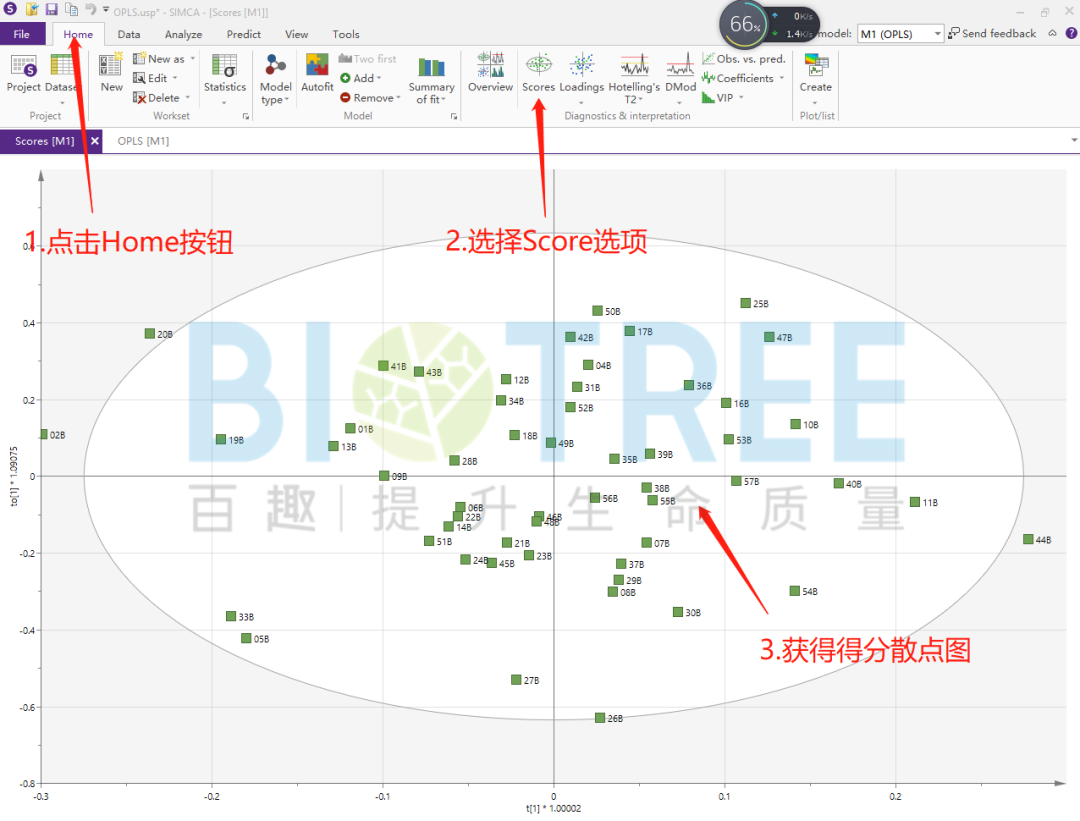
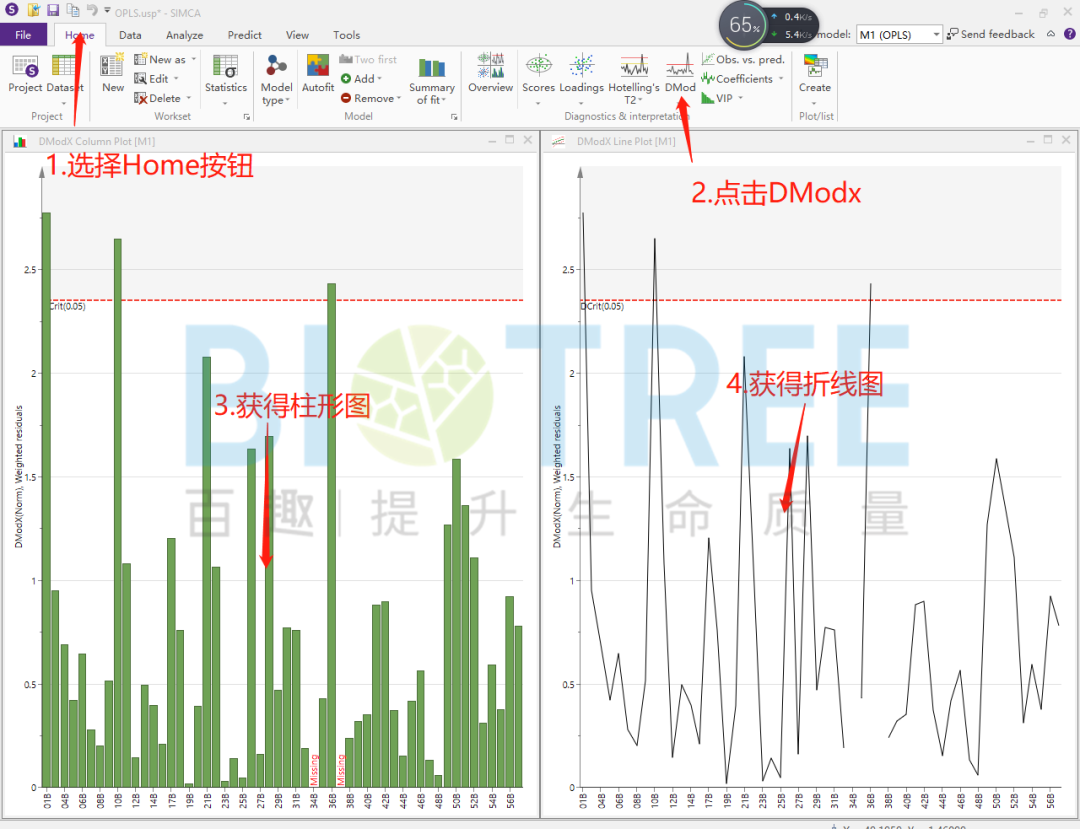 得分散点图和DModx图可考察样本中是否有异常样本点,当样本点距离95%置信区间过远和远远大于红线时可以去除该样本。
得分散点图和DModx图可考察样本中是否有异常样本点,当样本点距离95%置信区间过远和远远大于红线时可以去除该样本。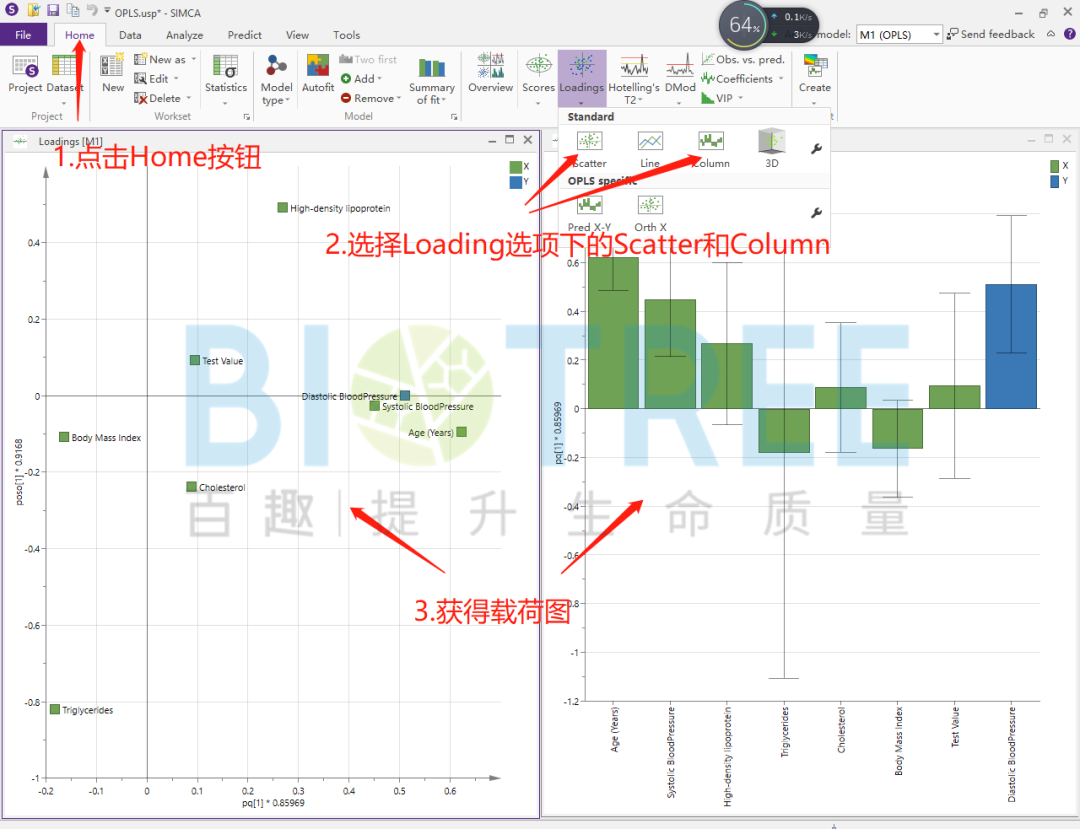
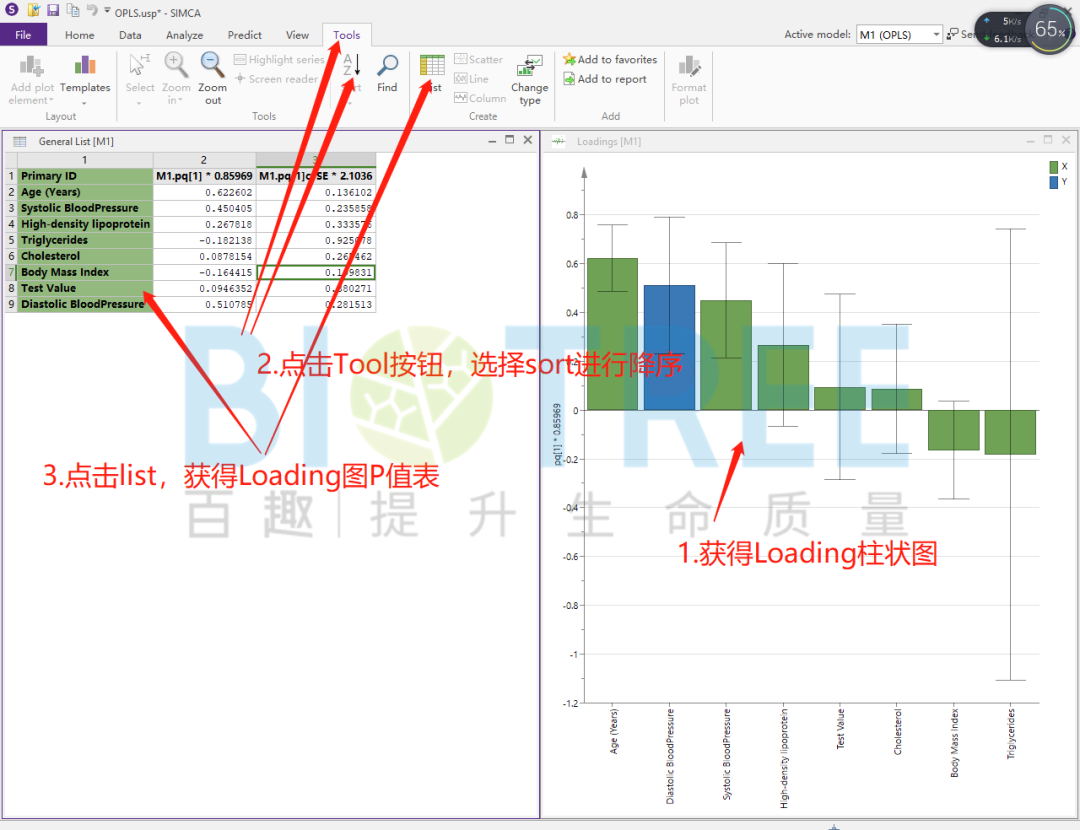
载荷图结合其柱形图可直观考察其他X变量和Y变量的相关性程度。
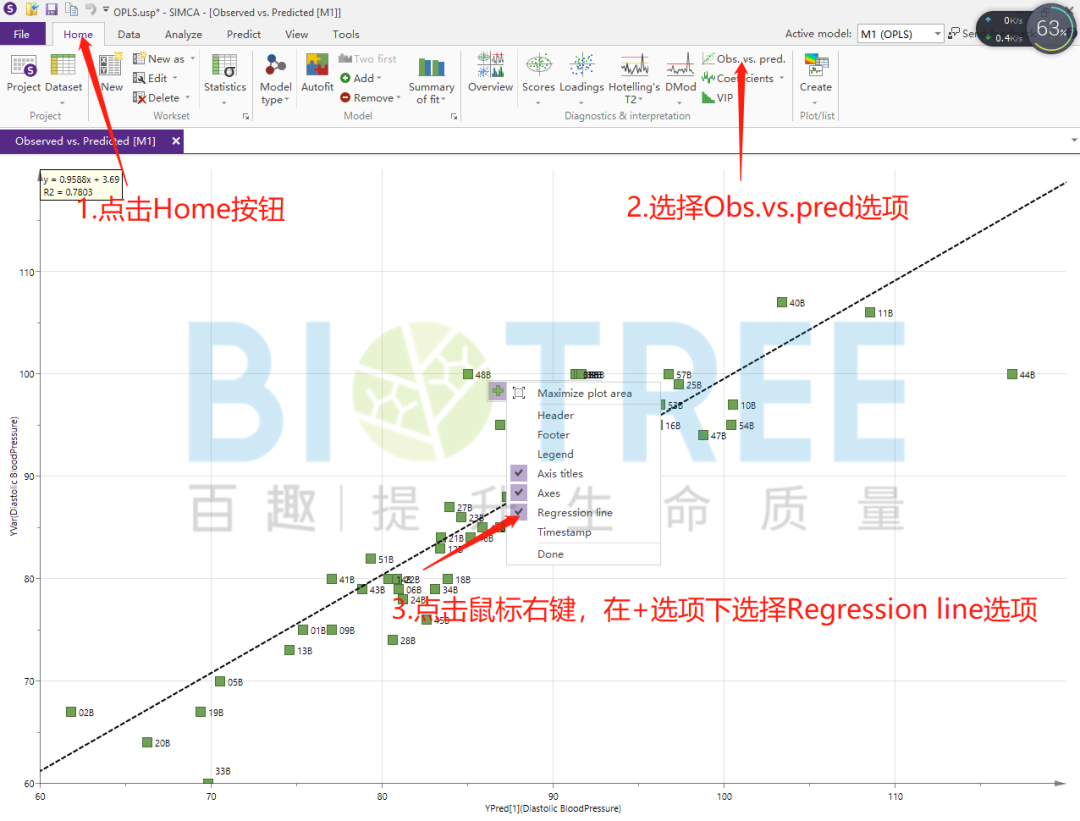
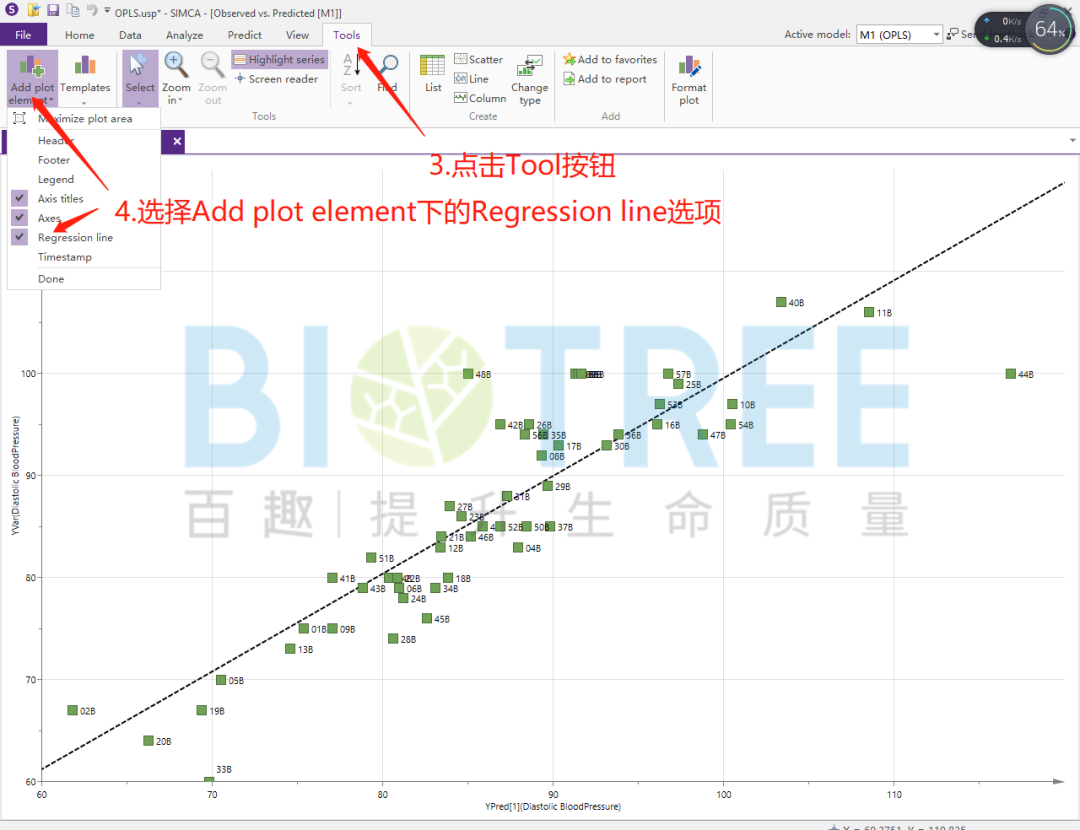 关于Y值和预测Y值的回归分析,可考察是否有异常样本点,以及模型的拟合程度。
关于Y值和预测Y值的回归分析,可考察是否有异常样本点,以及模型的拟合程度。
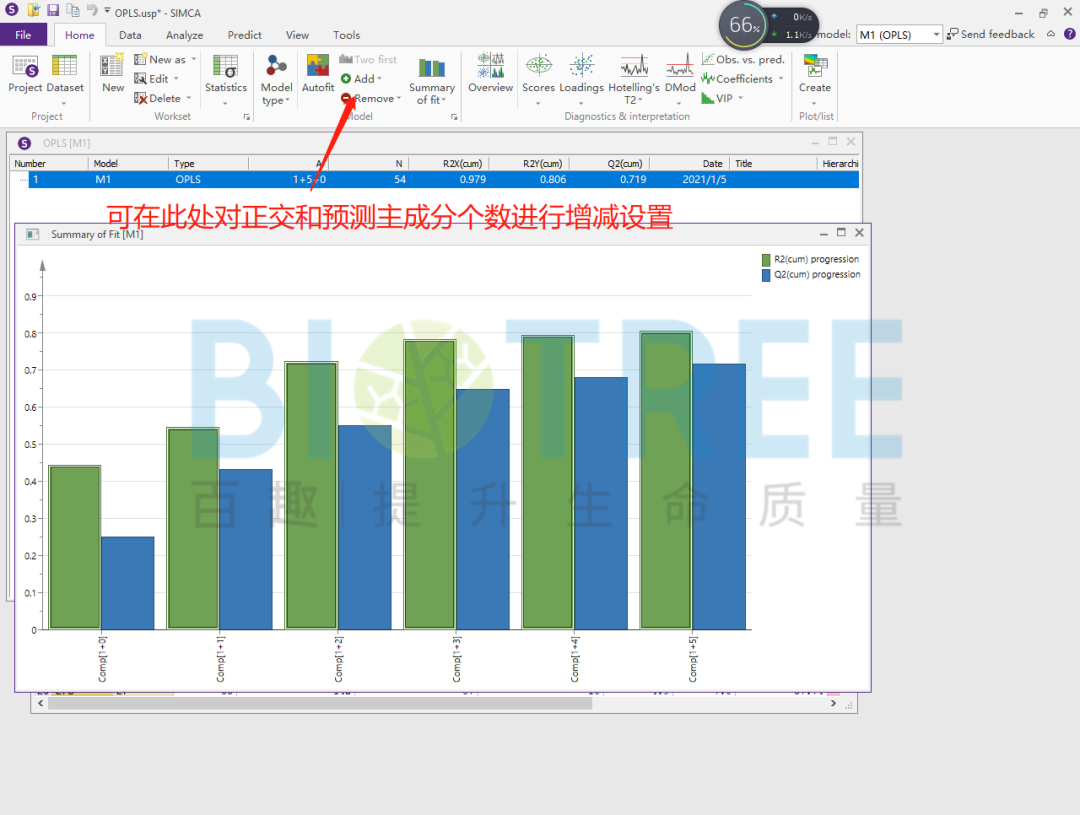
模型合格的最低标准R2>0.3,Q2>0.5。随着主成分个数增加,会逐渐出现过度拟合的现象。
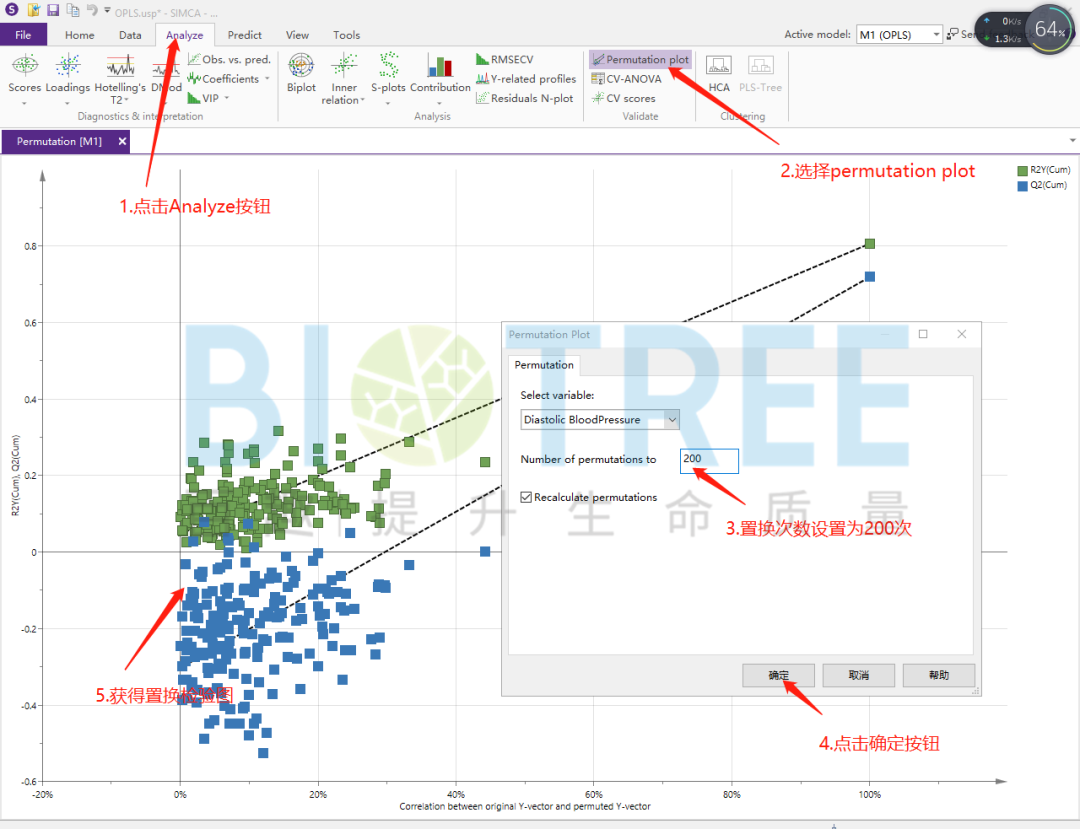
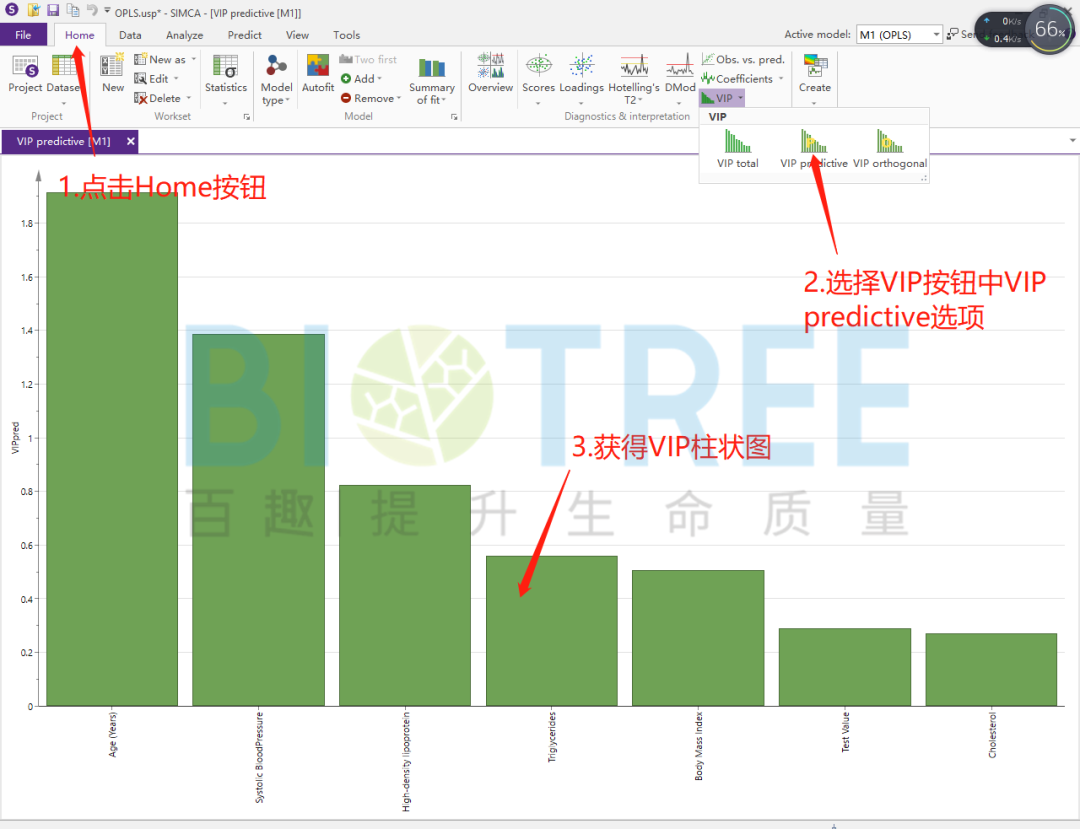
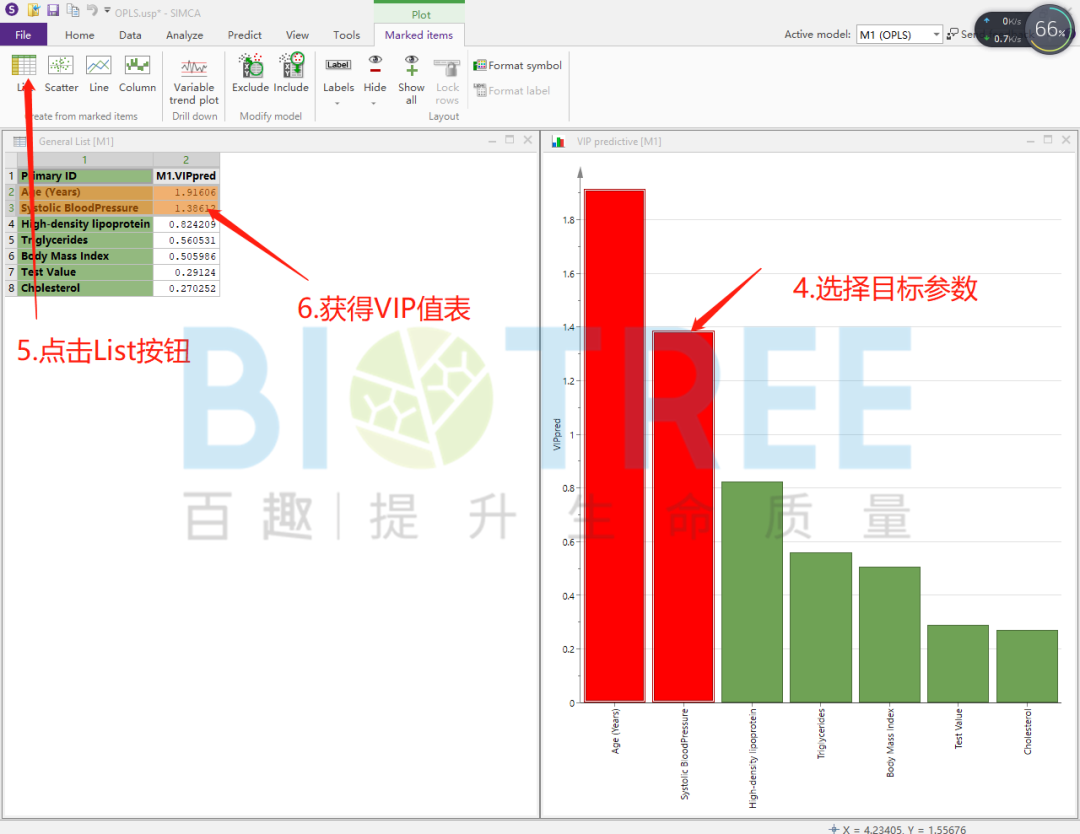
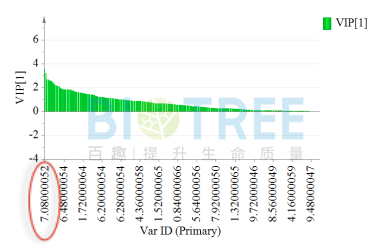
对关键X变量进行筛选,主要有三种方式VIP值排序、Loading图 P值以及S-plot图。
(1)常规数据分析
宏观数据或实验条件与代谢物数据之间的关联。对于功能、质量、等级等宏观数据或者时间、浓度等实验条件对于代谢物影响这类实验中,我们关注的是在不同自变量参数条件下有着显著变化的代谢物。OPLS-DA适用于两两之间的分析。可能这个时候,研究者不得不寻求其他的解决方案,比如说做ANOVA分析等。然而这些分析方法都是对代谢物进行逐一考察,缺乏整体层面上对代谢物重要性程度的考量,因此不能进一步告诉研究人员这些随自变量变化的代谢物中哪种代谢物更为关键。而OPLS回归分析,不仅适用于多个自变量参数设置这类场景,同时给出了代谢物的权重信息,因此可以筛选出不同自变量参数条件下最为重要的代谢物。
(2)数据挖掘
a.药效:从成分复杂的样本中寻找最关键的生物活性物质、快速解析物质功能;Mandrone Manuela等人[1]将胶原酶抑制活性设为参数Y,各种芳香族化合物设为变量X,通过OPLS回归分析中散点图,VIP值柱状图,寻找到了单宁类化合物是与胶原酶抑制活性最相关的物质。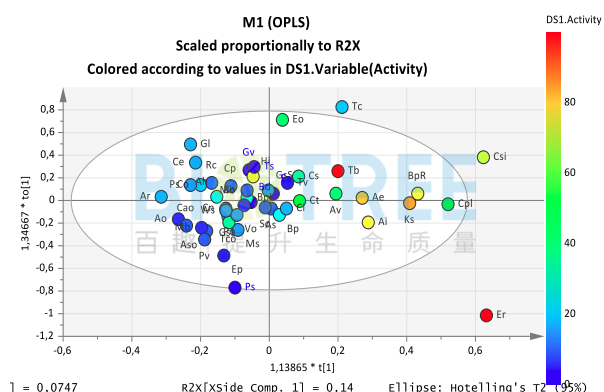
b.质量评价:筛选出影响质量、感官等的关键代谢物,获得评级和优化的线索;Yujing Zhang等人[2]在文章中将抗氧化能力设为参数Y。并将24个样本作为训练集建立了抗氧化能力和代谢物的OPLS回归模型,又将7个样本作为验证集测试,发现回归模型具有较好的对抗氧化能力的预测能力。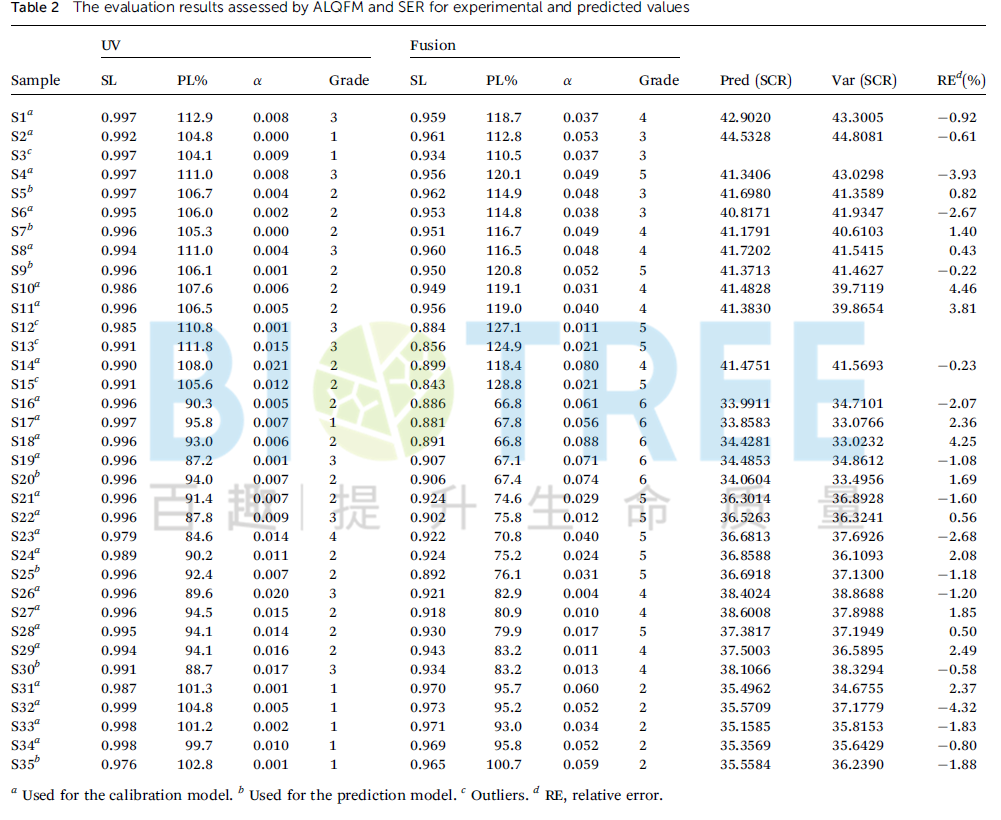
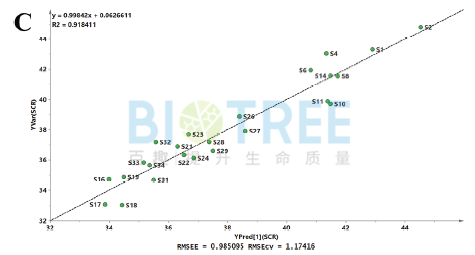
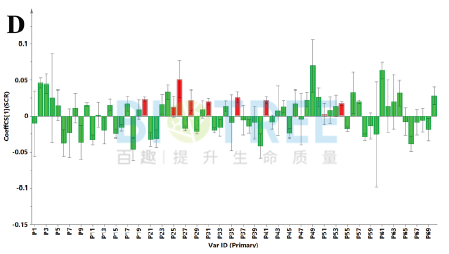 c.生产:分析不同产量、质量生产过程,明确关键生产条件和优化策略。Katsuaki Nitta等人[3]将丁醇产量设置为变量Y和 大肠杆菌内代谢物设置为变量X,并进行OPLS回归分析,其中乙酰辅酶A的回归系数最大,蛋氨酸的回归系数最小,然后基于这两种代谢物对大肠杆菌丁醇产量进行优化。
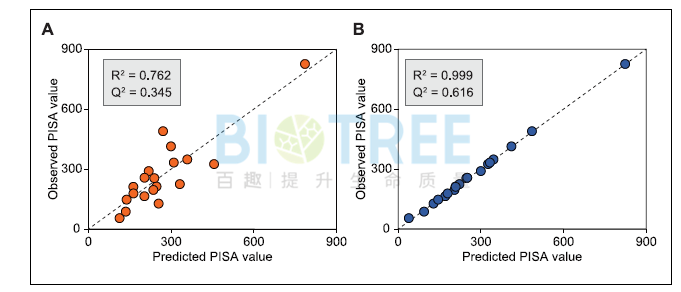
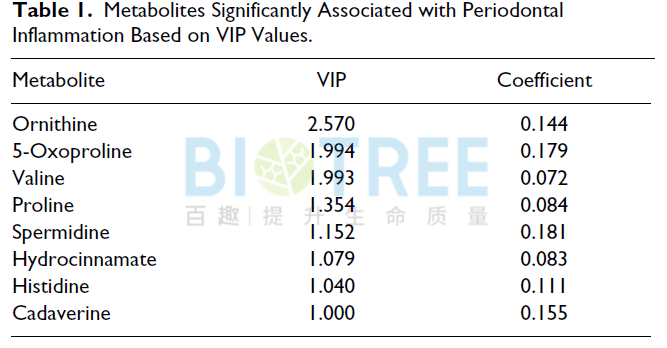
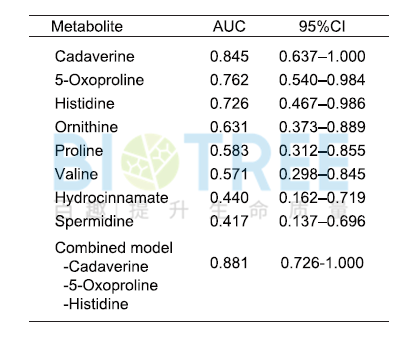
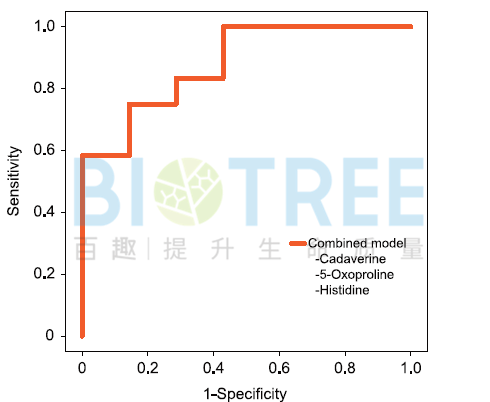
c.生产:分析不同产量、质量生产过程,明确关键生产条件和优化策略。Katsuaki Nitta等人[3]将丁醇产量设置为变量Y和 大肠杆菌内代谢物设置为变量X,并进行OPLS回归分析,其中乙酰辅酶A的回归系数最大,蛋氨酸的回归系数最小,然后基于这两种代谢物对大肠杆菌丁醇产量进行优化。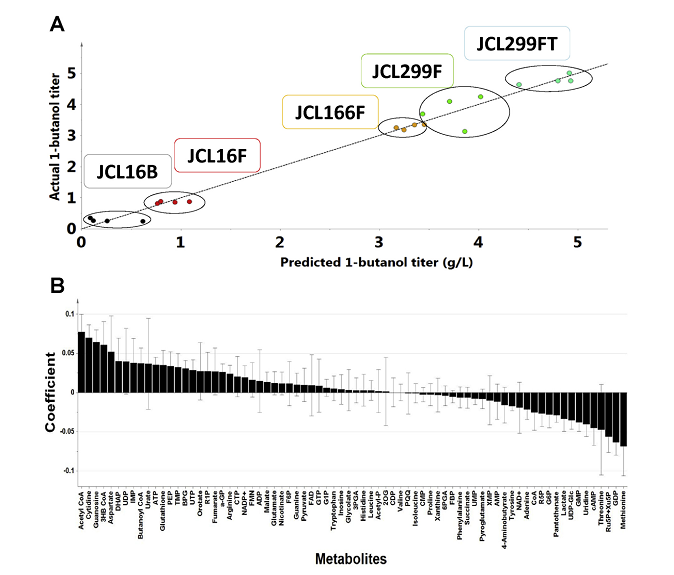 d.临床诊断:分析发病过程中趋势性变化的代谢物,获得潜在的诊断标志物或治疗靶点;M. Kuboniwa等人[4]将临床参数PISA设置为变量Y,代谢物数据设置为X变量,并建立OPLS回归模型。通过VIP值和回归系数筛选到8种显著的差异代谢物。然后进一步通过ROC分析,确定5-氧代脯氨酸、组氨酸和尸胺为最优的诊断标志物。
d.临床诊断:分析发病过程中趋势性变化的代谢物,获得潜在的诊断标志物或治疗靶点;M. Kuboniwa等人[4]将临床参数PISA设置为变量Y,代谢物数据设置为X变量,并建立OPLS回归模型。通过VIP值和回归系数筛选到8种显著的差异代谢物。然后进一步通过ROC分析,确定5-氧代脯氨酸、组氨酸和尸胺为最优的诊断标志物。
今天SIMCA16关于OPLS回归分析小技能就分享到这里啦,OPLS回归分析对于功能相关的差异变量的筛选在食品、医疗、药物研发等领域的应用相当广泛,这里就不一一列举了。下期我们将进入分类分析PCA-Class的精彩世界,敬请期待。我们下期再见。
参考文献:
1. Mandrone, Manuela. Medicinal Plants from Ancient Tradition as a Source for Matrix Proteases Inhibitors. Study of Correlation between Biological Activity and Phytochemical Profile. Alma Mater Studiorum Università di Bologna.2016. DOI 10.6092/unibo/amsdottorato/7613.
2. Yujing Z , Chao W , Fangliang Y , et al. UHPLC-ESI-Q-TOF-MS/MS analysis, antioxidant activity combined fingerprints for quality consistency evaluation of compound liquorice tablets[J]. RSC Advances, 2018, 8(49):27661-27673.
3. Nitta K , Lavi A W A , Pontrelli S , et al. Orthogonal partial least squares /projections to latent structures regression-based metabolomics approach for identification of gene targets for improvement of 1-butanol production in Escherichia coli[J]. Journal of Bioscience & Bioengineering, 2017:S 13891 72317303079.
4. Kuboniwa M , Sakanaka A , Hashino E , et al. Prediction of Periodontal Inflammation via metabolic Profiling of Saliva[J]. Journal of Dental Research, 2016:1381.


